韓연구진이 개발한 꿈의 DNA 저장 기술, 데이터센터 통째로 담는다
GIST, 차세대 DNA 저장 기술 개발…"꿈의 저장 기술 현실로"
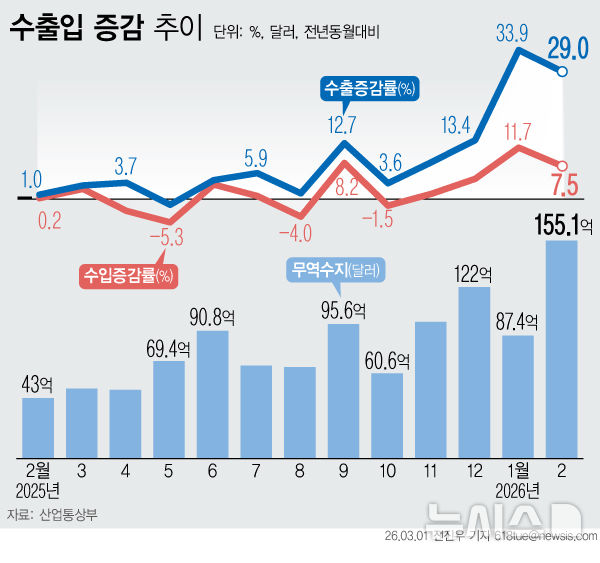
![[광주=뉴시스]순환적 DNA 합성 및 선택 기술의 개요.](https://img1.newsis.com/2025/02/25/NISI20250225_0001777707_web.jpg?rnd=20250225101828)
[광주=뉴시스]순환적 DNA 합성 및 선택 기술의 개요.
[광주=뉴시스] 배상현 기자 = 광주과학기술원 최영재 교수 연구팀이 개발한 '순환적 DNA 합성 및 선택' 기술로 기존 실리콘 반도체 메모리 한계를 뛰어넘는 대용량 데이터 저장이 가능해졌다.
광주과학기술원(GIST)은 신소재공학과 최영재 교수 연구팀이 서울대학교 권성훈 교수, ㈜에이티지라이프텍 연구팀과 공동으로 ‘순환적 DNA(디옥시리보핵산) 합성 및 선택’ 방식을 개발했다고 25일 밝혔다.
순환적 DNA 합성 및 선택방식은 특정 DNA 서열을 선택하기 위해 DNA 합성과 선택 과정을 반복적으로 수행하는 방법이다. 먼저 DNA 합성 단계에서 단일 나선 주형 DNA 위에서 목표 서열을 포함하는 DNA만 계속 합성되도록 조절하고, 그렇지 않은 서열은 멈추게 한다.
이후 선택 과정에서 원하는 서열만 남기고 나머지는 제거하며, 이 과정을 여러 번 반복해 최종적으로 특정 DNA 서열만 남긴다. 이를 통해 프라이머 없이도 원하는 DNA 서열을 정밀하게 선택할 수 있다.
연구팀이 개발한 ‘순환적 DNA 합성 및 선택’ 기술은 단일 염기 수준의 바코드를 활용해 프라이머 없이도 DNA 파일을 계층 구조로 탐색할 수 있도록 설계됐다.
이는 컴퓨터의 폴더 탐색 방식과 유사한 개념으로, 기존 PCR(중합효수 연쇄반응) 방식 대비 비용이 10배 절감되고 접근 효율은 3배 이상 향상됐다.
![[광주=뉴시스]왼쪽부터 최영재 지스트 신소재공학과 교수, 김우진 석박통합과정 학생.](https://img1.newsis.com/2025/02/25/NISI20250225_0001777708_web.jpg?rnd=20250225101932)
[광주=뉴시스]왼쪽부터 최영재 지스트 신소재공학과 교수, 김우진 석박통합과정 학생.
또 구분할 수 있는 DNA 파일의 수가 최소 7400만 배 이상 증가했으며, 특정 DNA 파일을 제거하고 새로운 DNA 파일을 삽입하는 등 파일 교체도 가능해졌다.
기존 PCR 방식에서는 DNA 파일마다 별도의 프라이머 한 쌍을 설계하고 합성해야 했지만,이 기술은 4개의 염기만으로 특정 DNA 파일에 접근할 수 있다.
이때 염기의 개수를 추가하는 방식으로 이론적으로 4n 개의 DNA 파일을 구분할 수 있어 확장성이 뛰어나다.
예를 들어 4개 염기의 바코드는 256종의 DNA 파일을, 8개 염기의 바코드는 6만5536종의 DNA 파일을 구분할 수 있다. 또한 일반적인 프라이머의 길이인 20개의 염기를 이용하면 이론적으로 4150억 개의 하위 집합을 인코딩할 수 있다.
최영재 교수는 “이번 연구를 통해 프라이머 없이도 특정 DNA 파일에 접근할 수 있는 새로운 방법론을 제시해 계층적 바코드 시스템을 적용함으로써 기존 PCR 방식의 한계를 극복했다”고 설명했다.
최 교수는 “향후 바코드 설계 최적화 및 자동화된 시스템과 결합하면 차세대 DNA 파일 접근 기술로서 DNA 기반 저장 시스템 상용화에 중요한 돌파구가 될 것으로 기대한다”고 밝혔다.
이번 연구는 최 교수가 지도하고 김우진 석박통합과정생과 고윤혜 석사과정생이 함께 수행했으며, 과학기술정보통신부 STEAM연구사업(미래유망융합기술파이오니어)의 지원을 받았다. 연구 결과는 국제학술지 `네이처 커뮤니케이션즈(Nature Communications)'에 지난 12일 온라인 게재됐다.
◎공감언론 뉴시스 [email protected]
Copyright © NEWSIS.COM, 무단 전재 및 재배포 금지